Manipulación de dataframes con dplyr
Resumen
Enseñando: 30 min
Ejercicios: 10 minPreguntas
¿Cómo puedo manipular los dataframe sin repetirme?
Objectivos
Ser capaz de utilizar los 6 principales ‘verbos’ para la manipulación de dataframes con pipes en
dplyr.Entender cómo
group_by()ysummarize()pueden combinarse para resumir datasets.Ser capaz de analizar un subconjunto de datos usando filtros lógicos.
La manipulación de dataframes significa muchas cosas para los investigadores, solemos seleccionar varias observaciones (filas) o variables (columnas), nosotros siempre agrupamos los datos por ciertas variable(s), o si incluso calculamos estadísticas de resumen. Podemos hacer esas operaciones usando operaciones de R base normal.
mean(gapminder[gapminder$continent == "Africa", "gdpPercap"])
[1] 2193.755
mean(gapminder[gapminder$continent == "Americas", "gdpPercap"])
[1] 7136.11
mean(gapminder[gapminder$continent == "Asia", "gdpPercap"])
[1] 7902.15
Pero esto no es muy eficiente, y puede volverse tedioso rapidamente porque hay bastante repetición. Repetirte a ti misma te va a costar tiempo, ahora y luego, y potencialmente puede introducir algunos errores.
El paquete dplyr
Afortunadamente, el paquete dplyr provee un
número muy útil de funciones para manipular dataframes en una forma que
reduce la repetición antes mencionada, asi como la probabilidad de cometer errores y posiblemente también te ahorre cierto tipeo. Como bonus, vas a encontrar la gramática de dplyr incluso más fácil de leer.
Vamos a cubrir 6 de las funciones más comunes usando pipes (%>%) para combinarlas.
select()filter()group_by()summarize()mutate()
Si aún no tienes instalado este paquete, por favor hazlo ahora:
install.packages('dplyr')
Ahora vamos a cargar el paquete:
library("dplyr")
Usando select()
Si, por ejemplo, queremos seguir solo con unas pocas de las variables en nuestro dataframe, podemos usar la función select(). Esto conservara solo las variables que seleccionas.
year_country_gdp <- select(gapminder, year, country, gdpPercap)
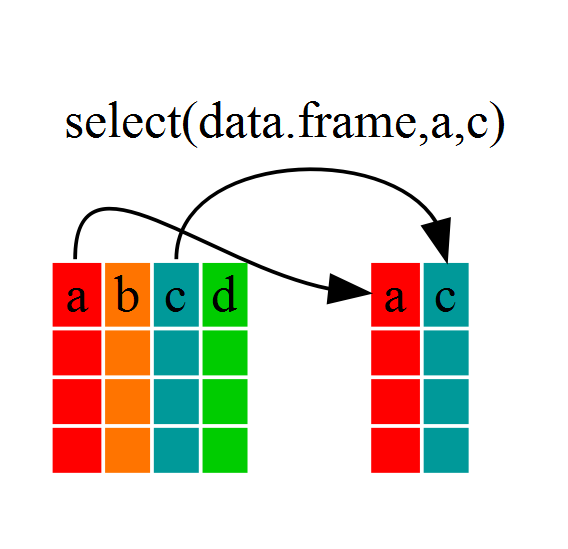
Si nosotros abrimos year_country_gdp vamos a ver que solo contiene año, país y gdpPercap. Hasta ahora hemos usado una gramática normal, pero las fortaleza de dplyr recae en combinar varias funciones usando pipes. Como la gramática de las pipes es diferente a todo lo que hemos visto en R hasta ahora, vamos a repertir que hemos hecho arriba usando pipes.
year_country_gdp <- gapminder %>% select(year,country,gdpPercap)
Para ayudarte a entender por qué lo escribimos en esa forma, vamos a recorrerlo paso
a paso. Primero convocamos a la base de datos gapminder y le pasamos, usando el
símbolo pipe %>%, al siguiente paso, que es la función select (). En este
caso no le especificamos que objeto de datos usamos en la función select () ya que
la obtiene del pipe anterior. Dato curioso: Puedes haberte encontrado
pipes antes en la terminal. En R, el símbolo de pipe es %>% mientras que en la terminal es
I pero el concepto es el mismo!
Usando filter()
Si ahora queremos avanzar con lo anterior, pero solo los países Europeos, podemos combinar select y filter
year_country_gdp_euro <- gapminder %>%
filter(continent == "Europe") %>%
select(year, country, gdpPercap)
Desafío 1
Escribe un solo comando (que puede abarcar varias líneas e incluir pipes) que producirá un marco de datos que tenga los valores africanos para
lifeExp,countryyyear, pero no para los otros continentes. ¿Cuántas filas tiene tu marco de datos y por qué?Solución al desafío 1
year_country_lifeExp_Africa <- gapminder %>% filter(continent=="Africa") %>% select(year,country,lifeExp)
Como la vez pasada, primero pasamos el marco de datos gapminder a la función filter(),
luego pasamos la versión filtrada del marco de datos gapminder a la
función select(). Nota: El order en las operaciones es muy importante en este
caso. Si utilizamos ‘select’ primero, filter no podría encontrar la variable continent ya que la habríamos removido en el paso anterior.
Usando group_by() y summarize()
Ahora, se suponía que debíamos reducir la repetición propensa a errores de lo que se puede hacer con base R,
pero hasta ahora no lo hemos hecho, ya que tendríamos que repetir lo anterior para cada continente. En lugar de filter(), que sólo pasará observaciones que cumplan con nuestros criterios (en lo anterior: continent=="Europe"),
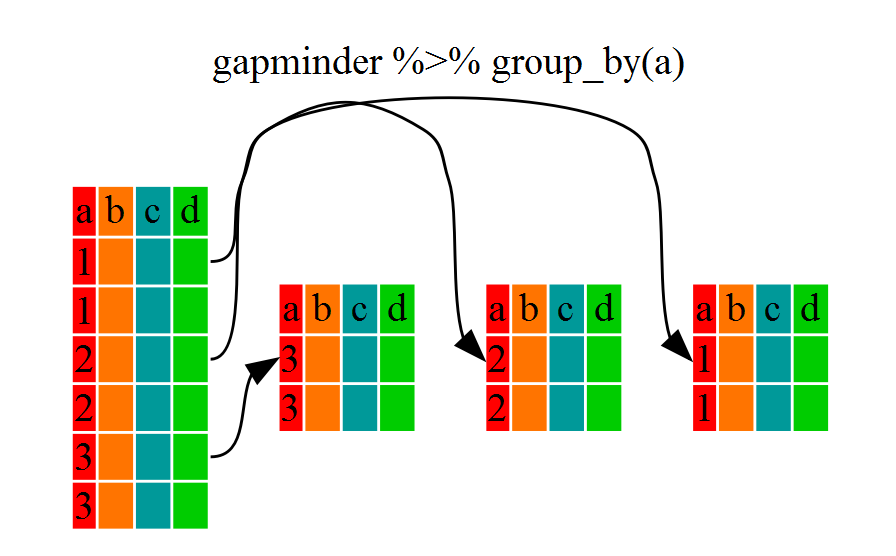
podemos usar group_by(), que esencialmente usará todos los criterios únicos que podrías haber usado en filter.
str(gapminder)
'data.frame':\t1704 obs. of 6 variables:
$ country : chr "Afghanistan" "Afghanistan" "Afghanistan" "Afghanistan" ...
$ year : int 1952 1957 1962 1967 1972 1977 1982 1987 1992 1997 ...
$ pop : num 8425333 9240934 10267083 11537966 13079460 ...
$ continent: chr "Asia" "Asia" "Asia" "Asia" ...
$ lifeExp : num 28.8 30.3 32 34 36.1 ...
$ gdpPercap: num 779 821 853 836 740 ...
gapminder %>% group_by(continent) %>% str()
tibble [1,704 × 6] (S3: grouped_df/tbl_df/tbl/data.frame)
$ country : chr [1:1704] "Afghanistan" "Afghanistan" "Afghanistan" "Afghanistan" ...
$ year : int [1:1704] 1952 1957 1962 1967 1972 1977 1982 1987 1992 1997 ...
$ pop : num [1:1704] 8425333 9240934 10267083 11537966 13079460 ...
$ continent: chr [1:1704] "Asia" "Asia" "Asia" "Asia" ...
$ lifeExp : num [1:1704] 28.8 30.3 32 34 36.1 ...
$ gdpPercap: num [1:1704] 779 821 853 836 740 ...
- attr(*, "groups")= tibble [5 × 2] (S3: tbl_df/tbl/data.frame)
..$ continent: chr [1:5] "Africa" "Americas" "Asia" "Europe" ...
..$ .rows : list<int> [1:5]
.. ..$ : int [1:624] 25 26 27 28 29 30 31 32 33 34 ...
.. ..$ : int [1:300] 49 50 51 52 53 54 55 56 57 58 ...
.. ..$ : int [1:396] 1 2 3 4 5 6 7 8 9 10 ...
.. ..$ : int [1:360] 13 14 15 16 17 18 19 20 21 22 ...
.. ..$ : int [1:24] 61 62 63 64 65 66 67 68 69 70 ...
.. ..@ ptype: int(0)
..- attr(*, ".drop")= logi TRUE
Notarás que la estructura del marco de datos donde usamos group_by()
(grouped_df) no es igual que el (data.frame) gapminder original.
grouped_df puede considerarse como una lista donde cada elemento de la lista es un
data.frame que contiene solo las filas que corresponden a un valor particular de continente (al menos en el ejemplo anterior).
Usando summarize()
Lo anterior fue sin complicaciones, pero group_by () es mucho más emocionante junto con summarize(). Esto nos permitirá crear nuevas variable(s)
mediante el uso de funciones que se repiten para cada marco de datos específico continente.
Es decir, usando la función group_by(), dividimos nuestro
marco de datos original en varias piezas, entonces podemos ejecutar las funciones
(por ejemplo, mean() o sd()) dentro de summarize().
(e.g. mean() or sd()) within.
gdp_bycontinents <- gapminder %>%
group_by(continent) %>%
summarize(mean_gdpPercap = mean(gdpPercap))
`summarise()` desgrupa el resultado (sobreescribe el argumento `.groups` )
gdp_bycontinents
# A tibble: 5 x 2
continent mean_gdpPercap
<chr> <dbl>
1 Africa 2194.
2 Americas 7136.
3 Asia 7902.
4 Europe 14469.
5 Oceania 18622.
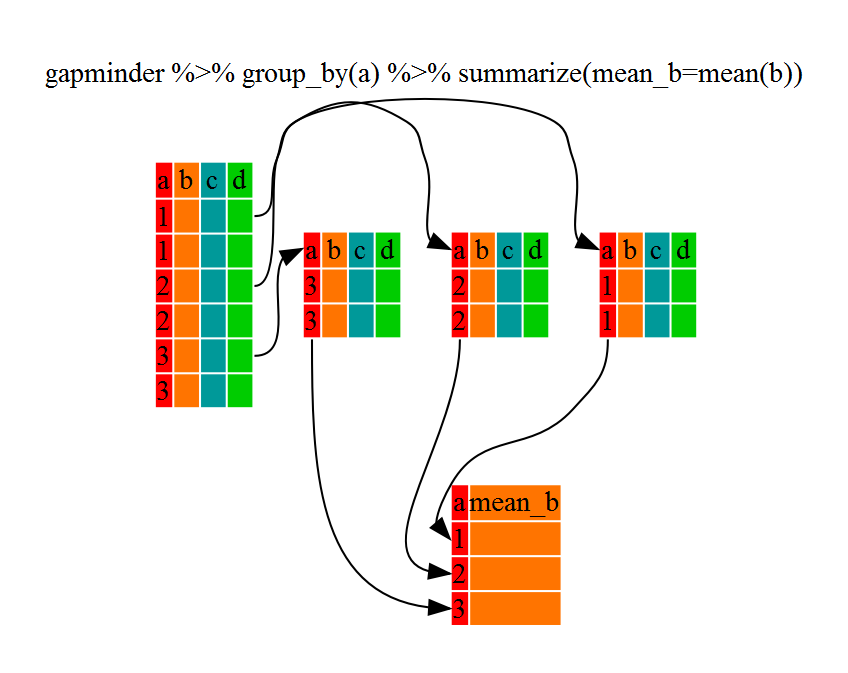
Eso nos permite calcular la media de gdpPercap para cada continente, pero esto se pone mejor aún.
Desafío 2
Calcula la esperanza de vida media por país. ¿Qué país tiene la esperanza de vida más larga and qué país tiene la esperanza de vida más corta?
Solución al Desafío 2
lifeExp_bycountry <- gapminder %>% group_by(country) %>% summarize(mean_lifeExp=mean(lifeExp))`summarise()` ungrouping output (override with `.groups` argument)lifeExp_bycountry %>% filter(mean_lifeExp == min(mean_lifeExp) | mean_lifeExp == max(mean_lifeExp))# A tibble: 2 x 2 country mean_lifeExp <chr> <dbl> 1 Iceland 76.5 2 Sierra Leone 36.8Otra manera de hacer esto, es usar del paquete
dplyrla funciónarrange(), que ordena las filas en un data frame según el orden de una o más variables del data frame. Tiene una sintaxis similar a otras funciones del paquetedplyr. Puedes usardesc()dentro dearrange()para ordenar en forma descendiente.lifeExp_bycountry %>% arrange(mean_lifeExp) %>% head(1)# A tibble: 1 x 2 country mean_lifeExp <chr> <dbl> 1 Sierra Leone 36.8lifeExp_bycountry %>% arrange(desc(mean_lifeExp)) %>% head(1)# A tibble: 1 x 2 country mean_lifeExp <chr> <dbl> 1 Iceland 76.5
La función group_by() nos permite agrupar por múltiples variables. Vamos a agrupar por year y continent.
gdp_bycontinents_byyear <- gapminder %>%
group_by(continent, year) %>%
summarize(mean_gdpPercap = mean(gdpPercap))
`summarise()` regrouping output by 'continent' (override with `.groups` argument)
Eso ya es bastante poderoso, ¡pero se pone aún mejor! No estás limitado a definir 1 nueva variable en summarize().
gdp_pop_bycontinents_byyear <- gapminder %>%
group_by(continent,year) %>%
summarize(mean_gdpPercap = mean(gdpPercap),
sd_gdpPercap = sd(gdpPercap),
mean_pop = mean(pop),
sd_pop = sd(pop))
`summarise()` regrouping output by 'continent' (override with `.groups` argument)
count() y n()
Una operación muy común es contar el número de observaciones para cada grupo.
El paquete dplyr tiene dos funciones relacionadas que ayudan con eso.
Por ejemplo, si quisiésemos verificar el number de países incluidos en el
dataset para el año 2002, podríamos usar la función count() . Toma los nombres
de una o más columnas que contienen los grupos que nos interesan, y opcionalmente
podemos ordenar de manera descendente los resultados al agregar sort=TRUE:
gapminder %>%
filter(year == 2002) %>%
count(continent, sort = TRUE)
continent n
1 Africa 52
2 Asia 33
3 Europe 30
4 Americas 25
5 Oceania 2
Si necesitamos utilizar el número de observaciones en cálculos, la función n()
es útil. Por ejemplo, si quisiésemos obtener el error estándar de la
esperanza de vida por continente:
gapminder %>%
group_by(continent) %>%
summarize(se_le = sd(lifeExp)/sqrt(n()))
`summarise()` desgrupa el resultado (sobreescribe el argumento `.groups` )
# A tibble: 5 x 2
continent se_le
<chr> <dbl>
1 Africa 0.366
2 Americas 0.540
3 Asia 0.596
4 Europe 0.286
5 Oceania 0.775
También puedes encadenar varias operaciones de resumen; en este caso calculando el minimum, maximum, mean and se (mínimo, máximo, promedio y error estándar) de la esperanza de vida por país para cada continente:
gapminder %>%
group_by(continent) %>%
summarize(
mean_le = mean(lifeExp),
min_le = min(lifeExp),
max_le = max(lifeExp),
se_le = sd(lifeExp)/sqrt(n()))
`summarise()` desgrupa el resultado (sobreescribe el argumento `.groups` )
# A tibble: 5 x 5
continent mean_le min_le max_le se_le
<chr> <dbl> <dbl> <dbl> <dbl>
1 Africa 48.9 23.6 76.4 0.366
2 Americas 64.7 37.6 80.7 0.540
3 Asia 60.1 28.8 82.6 0.596
4 Europe 71.9 43.6 81.8 0.286
5 Oceania 74.3 69.1 81.2 0.775
Usando mutate
También podemos crear nuevas variables previas a (o incluso luego) de resumir información usando mutate()
gdp_pop_bycontinents_byyear <- gapminder %>%
mutate(gdp_billion = gdpPercap*pop/10^9) %>%
group_by(continent, year) %>%
summarize(mean_gdpPercap = mean(gdpPercap),
sd_gdpPercap = sd(gdpPercap),
mean_pop = mean(pop),
sd_pop = sd(pop),
mean_gdp_billion = mean(gdp_billion),
sd_gdp_billion = sd(gdp_billion))
`summarise()` regrouping output by 'continent' (override with `.groups` argument)
Otros recursos geniales
- R para Ciencia de Datos
- Hoja de Referencia: Domar Datos
- Introducción a dplyr
- Domar datos “data wrangling” con R y RStudio - Link en Inglés
Puntos Clave
Usar el paquete
dplyrpara manipular dataframes.Usar
select()para elegir variables dentro de un dataframe.Usar
filter()to elegir datos basados en valores.Usar
group_by()ysummarize()para trabajar con subconjuntos de datos.Usar
mutate()para crear nuevas variables.